Ученые, Федерального научно-клинического центра (ФНКЦ) физико-химической медицины и МФТИ разработали алгоритм, который позволяет быстро сравнивать между собой совокупности ДНК микроорганизмов, обитающих в разных средах. Программа поможет в разработке средств персонализированной медицины. Статья опубликована в журнале Bioinformatics.
Программный инструмент под названием MetaFast позволяет провести быстрый сравнительный анализ большого количества метагеномов. При исследовании микрофлоры кишечника пациентов могут быть выявлены микроорганизмы, ассоциируемые с каким-либо заболеванием или предрасположенностью к нему. Иными словами, с помощью программы можно проводить нецелевой экспресс-анализ маркеров, указывающих на наличие каких-либо заболеваний.
Затем, используя целевые методики, например, ПЦР (полимеразная цепная реакция — способ многократного копирования фрагментов ДНК), подтверждать и корректировать результаты. По словам ученых, программа потенциально способна сократить сроки создания новых лекарств в несколько раз.
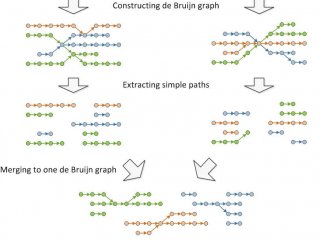
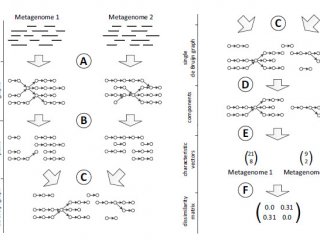
Одна из ключевых особенностей программы — возможность успешно работать со средами, генетическое содержимое которых ранее не было изучено. «Разработанный подход позволяет сделать сразу две вещи: найти все возможные генетические последовательности, даже если о них не было известно до этого (программа собирает их из кусочков геномных прочтений), и одновременно выделить метагеномные закономерности, которые отделяют одних пациентов от других. Например, людей с заболеванием и без», – отметил руководитель проекта Дмитрий Алексеев.
Диапазон применения MetaFast не ограничивается обнаружением патогенных микроорганизмов. К примеру, сравнение метагеномов самобытных народов в замкнутых популяциях с жителями городов может помочь выделить бактериальные штаммы, крайне полезные для людей, но утерянные в процессе урбанизации.